Resumen: La vida tal como la conocemos depende de la acción de 20 unidades constructivas proteicas, o aminoácidos. Mediante la adición de unos cuantos nuevos candidatos, los científicos esperan encontrar porqué ciertos organismos pueden ser capaces de apartarse tan ligeramente del gran Árbol de la Vida.
basado en un informe del Scripps Research Institute
Un equipo de investigadores del Instituto de Investigación Scripps y su Instituto Skaggs para Biología Química en La Jolla, California, ha modificado una línea de la bacteria
Escherichia coli para que utilice un código genético para 22 aminoácidos.

“... alguna pequeña charca caliente, con todos los tipos de sales amoniacales y fosfóricas, luz, calor, electricidad, etc ...”, Charles Darwin, sobre los orígenes de la vida en las charcas de marea.
Crédito: Smithsonian
|
'Hemos conseguido la incorporación simultánea de dos aminoácidos no naturales en el mismo polipéptido”, dice el Profesor Peter G. Schultz, titular de la cátedra de química del Scripps. “Ahora sabemos que el código genético es susceptible de ser ampliado a 22 aminoácidos, la siguiente cuestión es ¿hasta donde podemos llevarlo?”.
En una próxima entrega de la revista
Proceedings of the National Academy of Sciences, el equipo describe como han construido esta forma modificada de
E.coli para fabricar mioglobina con 22 aminoácidos, incorporando los no naturales O-methyl-L-tirosina y L-homoglutamina además de los 20 naturales.
La biomolécula, DNA, que se pliega en el interior del núcleo celular.
|
Los científicos han creado durante años proteínas con esos aminoácidos en el laboratorio, pero hasta que, varios años atrás, Schultz y sus colaboradores comenzaron su trabajo en este campo, nadie había dado con la manera de obligar a un organismo a añadir estos aminoácidos no naturales en su código genético. Los estudios preliminares del grupo de Schultz describían la incorporación simple de algunos aminoácidos no naturales útiles en química y biología, en
E. Coli y en la levadura
Saccharomyces cerevisiae.
Este último resultado es de gran ayuda porque demuestra que se pueden añadir múltiples aminoácidos no naturales al código genético de un único organismo modificado. Esta demostración abre la puerta para la fabricación de proteínas en el contexto de la célula viva con tres, cuatro o más aminoácidos adicionales al tiempo.
El artículo, “Un bacteria de 22 aminoácidos con un codón cuadruplete funcional” ('A twenty-two amino acid bacterium with a functional quadruplet codon') cuyos autores son J. Christopher Anderson, Ning Wu, Stephen W. Santoro, Vishva Lakshman, David S. King, and Peter G. Schultz será publicado electrónicamente durante la semana del 10 al 16 de Mayo por la revista Proceedings of the National Academy of Sciences. El artículo aparecerá impreso más tarde este año.
Este trabajo ha sido financiado por el Departamento de Energía y el Instituto Skaggs. Los científicos involucrados en este estudio fueron respaldados por una beca predoctoral de la Fundación Nacional para la Ciencia (National Science Foundation), una beca del Instituto canadiense de investigación de la Salud (Canadian Institutes of Health Research) y un Premio de Carrera en Ciencias Biomédicas del Fundación Burroughs Wellcome.
¿Porqué expandir el Código Genético?
La vida tal como la conocemos se compone, a nivel molecular, de los mismos boques estructurales básicos. Por ejemplo, todas las formas de vida en la Tierra utilizan los mismos cuatro nucleótidos para fabricar ADN. Y con pocas excepciones, todas las formas de vida conocidas usan los mismos 20 aminoácidos, y sólo esos 20, para fabricar proteínas
La cuestión es porqué la vida se paró en 20, y porqué esos 20 en particular.
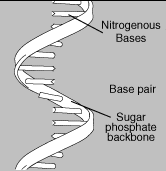
El ARN transporta el mensaje genético del ADN al citoplasma de la célula donde se fabrican las proteínas.
Crédito: Darryl Leja/Access Excellence.
|
Aunque la respuesta a esta pregunta pueda ser elusiva, la barrera de 20 aminoácidos no es absoluta. En algunos casos raros, de hecho, ciertos organismos han evolucionado la capacidad de utilizar los inusuales aminoácidos selenocisteína y pirrolisina, ligeras modificaciones de los aminoácidos cisteína y lisina.
Dejando aparte estas raras excepciones, los científicos han buscado a menudo formas de incorporar aminoácidos no naturales en proteínas tanto en tubo de ensayo como en el contexto de células vivientes, puesto que estas nuevas proteínas son de gran utilidad para la investigación biomédica básica. Proporcionan una poderosa herramienta para estudiar controlar los procesos biológicos que constituyen la base de algunos de los más intrigantes problemas de la biofísica y la biología celular modernas, como la transducción de señales, el tráfico de proteínas en la célula, el plegamiento de proteínas, y las interacciones proteína-proteína.
Por ejemplo, hay aminoácidos nuevos que contienen grupos fluorescentes que pueden utilizarse para el marcaje de proteínas en sitios específicos con pequeñas etiquetas fluorescentes y observarlas
in vivo. Esto es particularmente útil ahora que se ha secuenciado el genoma humano y los científicos han puesto su atención en lo que estos genes están haciendo dentro de las células.
Otros aminoácidos no naturales contienen etiquetas fotoafines y otros “enlazantes” que se pueden utilizar para inmovilizar interacciones proteína-proteína forzando a las proteínas interaccionantes a ligarse covalentemente una con otra. La purificación de esas proteínas ligadas puede permitir a los científicos ver que proteínas interaccionan en las células vivas, incluso aquellas con interacciones débiles que son difíciles de detectar por los métodos actuales.
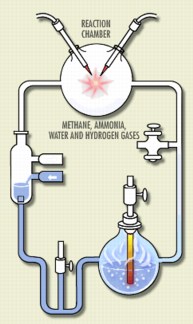
El experimento Miller-Urey generaba chispas eléctricas (simulando relámpagos) en una mezcla de gases diseñada para parecerse a la atmósfera primitiva de la Tierra.
Crédito: AccessExcellence.org
|
Los aminoácidos no naturales también son importantes en medicina, y muchas proteínas utilizadas terapéuticamente necesitan ser modificadas con grupos químicos como polímeros, agentes enlazantes, y moléculas citotóxicas. El año pasado, Shultz y sus colegas del Scripps demostraron también que se podían incorporar en sitios específicos aminoácidos glicosilados para producir proteínas glicosiladas, un paso importante en la preparación de algunos medicamentos.
Nuevos aminoácidos hidrofóbicos, aminoácidos ligantes de metales pesados y aminoácidos que contienen “etiquetas de spin” (spin labels) pueden resultar útiles para sondear la estructura de las proteínas en las que están insertados. Y aminoácidos no usuales que contienen características químicas como grupos “ceto”, que son como bloques de LEGO, pueden utilizarse para ligar otros compuestos químicos como moléculas de azúcar, que podrían ser relevantes en la producción de proteínas terapéuticas.
Combinando la supresión Ámbar con la supresión de cambio de marco
Schultz y sus colegas han tenido éxito en la fabricación de la
E. coli de 22 aminoácidos explotando la redundancia del código genético. Cuando se expresa una proteína un enzima lee las bases del DNA de un gen (A, G, C, y T), y las transcribe en ARN (A, G, C, y U). Este denominado “ARN mensajero” (ARNm) se traduce mediante otro complejo proteína-ARN (el ribosoma) en una proteína. El ribosoma requiere la colaboración de moléculas de ARN transferente (ARNt) las cuales tienen “cargado” un aminoácido, para lo que requieren de una enzima “cargadora”.
Cada ARNt reconoce una combinación específica de tres bases, el “codón”, en el ARNm y está cargado con un único aminoácido que es específico para ese codón.
Durante la síntesis de proteínas, el ARNt específico para el siguiente codón en el ARNm viene cargado con el aminoácido correcto, y el ribosoma coge el aminoácido y lo incorpora a la cadena proteica creciente.
La redundancia del código genético procede del hecho de que hay más codones que aminoácidos utilizados. De hecho, hay 4x4x4=64 posibles maneras deiferentes de tener un codón, o cualquier combinación de cuatro letras tomadas de tres en tres en el ARNm (UAG, ACG, UCC, etc.). Con sólo 20 aminoácidos utilizados por los organismos, teóricamente no todos los codones son necesarios.

Síntesis de la vida extrema:
• Más calor: (113º C) Pyrolobus fumarii (Isla Vulcano, Italia)
• Más frío: (-15º C) Cryptoendoliths (Antártida)
• Radiación más alta: (5 MRad, o 5000 veces la radiación letal para los humanos) Deinococcus radiodurans
• Mayor profundidad: 3.2 km bajo el suelo
• Mayor acidez: pH 0.0 (La mayoría de la vida está a un factor al menos 100,000 veces menos ácido)
• Mayor basicidad: pH 12.8 (La mayoría de la vida está a un factor al menos 1000 veces menos básico)
• Mayor duración en el espacio: (6 años) Bacillus subtilis (en un satélite de la NASA )
• Mayor presión: (1200 veces la atmosférica)
• Mayor salinidad: (30% sal, o 9 veces la salinidad de la sangre humana). Haloarcula
• Menor tamaño: (<0.1 micras o 500 veces menor que el grosor de un cabello humano (picoplancton)
Crédito: USGS
|
Sin embargo la naturaleza los utiliza de todas formas. Algunos de los 64 codones son redundantes, codifican para el mismo aminoácido, y tres de ellos son codones sin sentido, no codifican para ningún aminoácido.
Estos codones sin sentido son muy útiles porque normalmente cuando un ribosoma que está sintetizando una proteína llega a uno de ellos, se disocia del ARNm y la síntesis termina. De ahí que también se conozca a los codones sin sentido como codones de terminación (“stop” codons). Uno de estos, el codón
ambar (UAG), juega un importante papel en la investigación de Schultz.
Schultz y sus colegas sabían que si pudieran dotar a sus células con una molécula de ARNt que reconozca UAG y asimismo las dotaran con una sintetasa que cargara este ARNt con un aminoácido no natural, tendrían la forma de insertar en sitios específicos los aminoácidos no usuales en la proteína que ellos quisieran.
Necesitaban encontrar una pareja ARNt/sintetasa funcional y relacionada, que reaccionen entre sí pero no con las parejas endógenas de
E. coli. Para ello desarrollaron una metodología que seleccionara la especificidad de la sintetasa que acepta selectivamente aminoácidos no naturales.
Empezando con una pareja adicional ARNt/sintetasa del organismo
Methanococcus jannaschii, crearon una familia de células de
E. coli donde cada una codificaba una sintetasa mutada de
Methanococcus jannaschii, y cambiaron su especificidad de forma que reconociera el aminoácido no natural O-metil-L-tirosina.
Para hacer esto, idearon una selección positiva donde sólo las células que cargan el ARNt adicional con cualquier aminoácido sobreviven. Luego diseñaron una selección negativa donde cualquier célula que reconozca UAG utilizando un ARNt cargado con un aminoácido diferente de la O-metil-L-tirosina moriría.
Haciendo esto encontraron la bacteria con la sintetasa extraña que carga el ARNt extraño con y únicamente el aminoácido no natural deseado. Cuando un ribosoma leyendo un ARNm en estas
E. coli encuentre un UAG insertará el aminoácido no natural O-metil-L-tirosina.
Es más, cualquier codón en un ARNm que se cambien a UAG codificará para el nuevo aminoácido en ese lugar, lo que da a Schultz y sus colegas una vía de incorporar nuevos aminoácidos en sitios especificos de las proteínas expresadas en
E. coli.
De forma parecida, Schultz y sus colaboradores construyeron una pareja artificial ARNt/sintetasa, procedente del organismo polar arcaico
Pyrococcus horikoshii, que reconoce el codón de cuatro bases AGGA.
El ARNt tiene un lazo anticodón de cuatro bases, y cuando un ribosoma leyendo un ARNm en las células de
E. coli encuentra AGGA inserta el aminoácido no natural L-homoglutamina en ese lugar.
Poniendo ambos sistemas en la misma célula de
E. coli, Schultz y sus colegas han demostrado, sin lugar a dudas, que es técnicamente posible tener sistemas mutuamente independientes operando al tiempo en la misma célula. Esto abre la posibilidad de hacer en el futuro múltiples sustituciones sencillas con otros aminoácidos no naturales.

